目的:通过此教程,了解Discovery Studio中基于蛋白序列构建同源跨膜蛋白模型的操作方法及结果分析。
所需功能和模块:Discovery Studio Client,DS Sequence Analysis,DS MODELER,DS Protein Families。
所需数据文件:2rh1.pdb,ADRB1_HUMAN.fasta。
所需时间:0.5小时
介绍
膜蛋白在药物研发领域中是一类非常重要的靶标蛋白,这是由于该类蛋白,尤其是GPCR(G蛋白偶联受体),是目前许多已知药物以及仍处于研发阶段的大量药物分子的目标靶点。GPCR是一类七次跨膜受体蛋白,它们广泛地参与细胞增殖、分化、迁移,尤其是各类生理活动的调控。从近几十年药物发展的历史看,全球70%所开发的药物都是针对GPCR的,因此GPCR是令人瞩目的药物治疗靶点。
本教程即演示了如何使用DS中已有的一系列工具(tools)和流程(protocols),以beta-2肾上腺素受体X-衍射晶体结构为模板,来构建beta-1肾上腺素受体的同源模型并进行加膜处理。
本教程包括以下步骤:
-
输入beta-1肾上腺素受体的序列及beta-2肾上腺素受体模板结构
-
预测蛋白的跨膜区
-
将目标序列比对至模板序列
-
构建3D同源模型
-
添加隐性生物膜至构建得到的模型
-
调整生物膜的位置
打开输入文件
在文件浏览器(Files Explorer)中,展开Samples | Tutorials | Protein Modeling文件夹,双击打开ADRB1_HUMAN.fasta序列文件。
这将在一个名称为ADRB1_HUMAN新的序列窗口中打开beta-1肾上腺素受体的一级序列(序列名为ADRB1_HUMAN)。
构建同源模型的第一步是识别合适的模板。如果同目标序列的序列一致性较高的模板存在,通常可以通过简单的BLAST搜索得到该模板结构。对于膜蛋白而言,X-单晶衍射解析得到的晶体结构非常有限,因此本教程不包含模板识别这一步骤(该步骤细节可以参看MODELLER教程)。
本教程采用最近解析出来的beta-2肾上腺素受体晶体结构(PDB号:2rh1)作为模板。
在文件浏览器(Files Explorer)中,展开Samples | Tutorials | Protein Modeling文件夹,双击打开2rh1.pdb文件。
蛋白2rh1.pdb在分子窗口中显示。
注:采用BLAST搜索PDB_nr95数据库可以找到另一个可能的模板,火鸡beta-1肾上腺素受体。该晶体结构同目标序列的序列一致性高达70.5%,是一个比较好的模板结构。然而,本教程采用2rh1作为模板是想突出DS的功能,因为在模板序列一致性如此高的情况下,软件功能在很大程度上是不太相关的。
建模的下一步:将目标序列与模板序列进行序列比对。首先需要将目标序列和模板序列置于同一个序列窗口当中。
点击激活ADRB1_HUMAN序列窗口。
在窗口中点击右键选择Insert Sequence | From Windows…
打开Insert Sequence from Windows对话框。
选择2rh1点击OK。
将2rh1序列插入ADRB1_HUMAN序列窗口当中。
注:此时两序列并未进行比对,状态栏(DS窗口左下角)显示了两序列之间的序列一致性及相似性情况(分别为7.0%,27.4%)。
预测蛋白的跨膜区
序列比对流程允许利用蛋白的二级结构信息来提高序列比对的精度。二级结构预测方法,如DSC,是基于球蛋白的溶剂暴露模式来定义的。膜蛋白的表面大部分都暴露于生物膜内部的脂质环境中而非水环境。这就导致了氨基酸序列中疏水残基和亲水残基模式的不同,从而使得标准的二级结构预测方法来预测膜蛋白的二级结构不太可靠。本教程则采用专门用于预测膜蛋白螺旋区的的TransMem方法。
在工具浏览器(Tools Explorer)中,展开Macromolecules | Analyze Transmembrane Proteins,点击Predict Transmembrane Helices。
在ADRB1_HUMAN序列窗口中相应地添加了蛋白二级结构卡通图,红色横条表示alpha-螺旋,蓝色箭头表示beta-折叠。(图1)
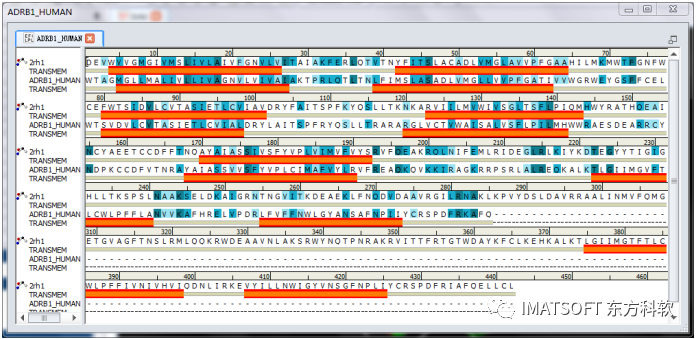
图1
注:预测2rh1的跨膜螺旋时,有一段区域(ASN1002—TYR1161)是没有预测到螺旋结构的。该区域对应的是一个为方便结晶与β-2肾上腺素受体融合的溶菌酶的序列。标准的二级结构预测方法可以预测出该区域的二级结构。如果将二级结构预测结果用于指导序列比对,如此大的序列插入会扰乱比对的结果。
将目标序列与模板序列进行比对
在工具浏览器(Tools Explorer)中,展开Macromolecules | Align Sequence and Structures,点击Align Sequences…打开Align Sequences对话框。
确保Input Sequence Set设置为ADRB1_HUMAN:All。
点击Use Secondary Structures右边的下拉菜单,选择TRANSMEM。(图2)
点击Run运行作业,等待作业完成。
该作业大概需要1.5min的时间(奔腾4,3GHz的CPU,1GB的存储器)。
作业完成以后,ADRB1_HUMAN序列窗口会自动更新为序列比对之后的结果(图3),同时弹出序列一致性和序列相似性(42.1%,53.3%)报告的对话框(图3)。
点击OK关闭该对话框。
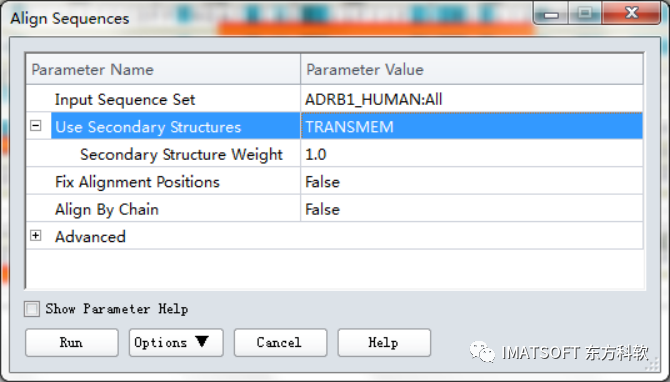
图2 “Align Sequences”参数设置
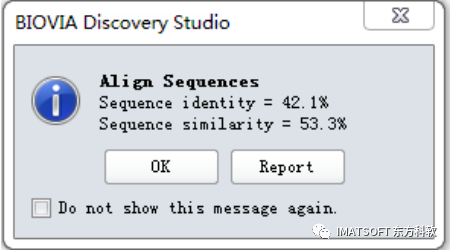
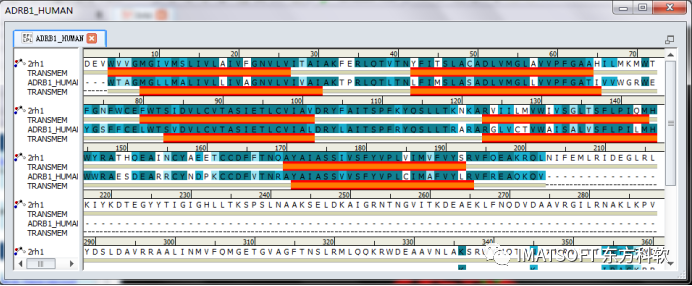
图3
构建目标序列的3D同源模型
在工具浏览器(Tools Explorer)中,展开Macromolecules | Create Homology Models,点击Build Homology Models,打开Build Homology Models对话框。
点击Input Sequence Alignment右边的栅格,下拉列表中选取ADRB1_HUMAN:All。
点击Input Templates Structures一栏,选择2rh1。
点击Input Model Sequence右边的栅格,下拉列表中选取ADRB1_HUMAN。
展开Copy From Templates参数栏,点击Ligands参数栏,选择2rh1:A:CLR412,2rh1:A:CLR413,2rh1:A:CLR414,2rh1:A:PLM415。
该步骤将棕榈酸和胆固醇分子从模板结构中直接复制到模型结构当中。这些分子有助于决定生物膜相对于模型结构的位置。
将Number of Models设为1。
点击Optimization Level右边的栅格,下拉列表中选取Low。(图4)
注:将Optimization Level由默认值改为Low,可以加快计算速度,但产生的模型的精度会下降。
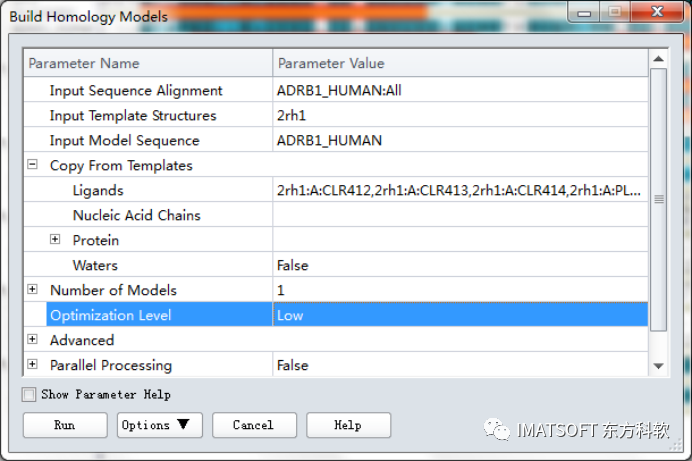
图4 “Build Homology Models”参数设置
点击Run运行该作业。
该作业大概需要1min的时间(奔腾4,3GHz的CPU,1GB的存储器)。
待作业完成以后,构建得到的模型在名为ADRB1_HUMAN的分子窗口中打开(图5),同时有一个报告PDF Total Energy的对话框弹出。
点击OK关闭该对话框。
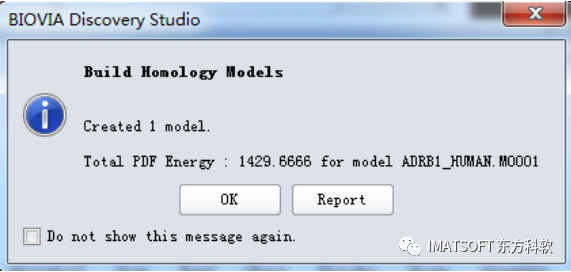
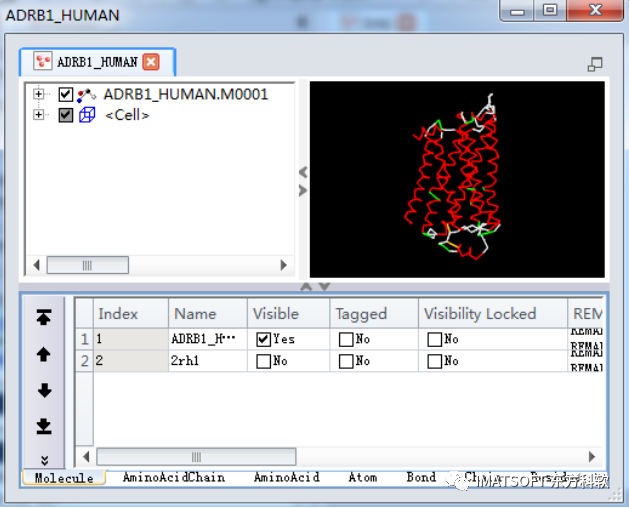
图5
给模型添加隐性生物膜结构
DS为用户提供了为蛋白质结构添加和控制隐性膜结构的工具。该膜结构的位置可以用在模拟(simulation)流程中以明确蛋白质在隐性溶剂模型中的溶剂化性质。
在菜单栏中,选择Edit | Preferences…以显示 Preferences 对话框。
在Transmembrane Protein 页面中, 确保Membrane Thickness的设置为30 Å(图6)。
点击OK关闭Preferences对话框。
这里所说的Membrane thickness指的是膜的疏水区,不包括磷脂的极性头部所占据的区域。

图6
点击并激活ADRB1_HUMAN分子窗口。
在工具浏览器(Tools Explorer)中,展开Macromolecules | Analyze Transmembrane Proteins,在Create and Edit Membrane栏下点击Add。
该步将为蛋白质结构添加一个膜结构。该膜在分子窗口的图形界面上以两个平行的平板表示(图7)。
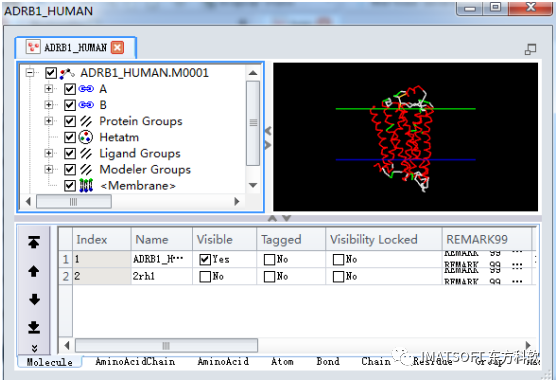
图7
调整膜的位置
Add命令只使用一个非常简单的溶剂模型来确定膜结构的位置。做这样的优化处理更多地是考虑到了速度而非精度。因此,通常都需要对膜的位置进行重新调整。
在系统(Hierarchy)视图中,展开ADRB1_HUMAN.M0001,点击选中B链以选中模型中的胆固醇和棕榈酸分子。
注意到,胆固醇和棕榈酸分子的极性区域都处于以两个平板所定义的膜结构的疏水区(图8)。这表明初始构建的膜的位置需要调整。
使用Add Membrane and Orient Molecule流程可以自动完成上述膜结构位置的调整过程。该protocol使用广义伯恩(GB)溶剂模型对膜的位置进行全面优化。由于此步计算需要非常长的计算时间,所以本教程使用Analyze Transmembrane Proteins工具面板来调整膜结构的位置。
在系统(Hierarchy)视图中,展开ADRB1_HUMAN.M0001,点击选中A链。
在工具浏览器(Tools Explorer)中,展开Macromolecules | Analyze Transmembrane Proteins,在Create and Edit Membrane栏下点击Add Orientation Monitor。
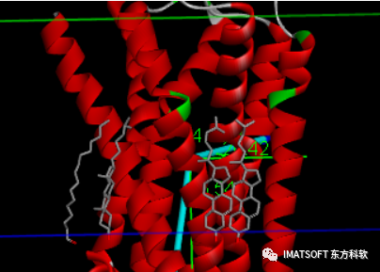
这将在模型结构窗口中添加一个膜取向调节坐标(membrane orientation monitor),用以指示膜与蛋白质主轴间的移动(shift),倾斜(tilt)及旋转(rotation)角度(图8)。
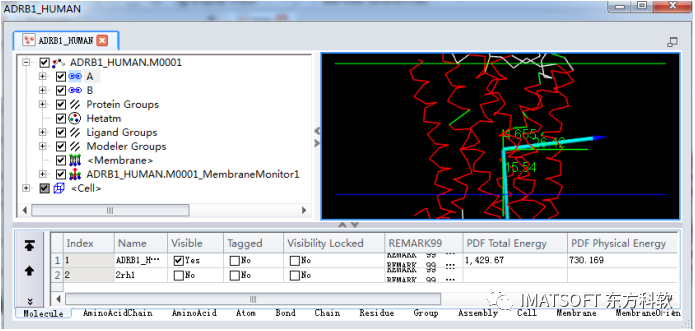
图8
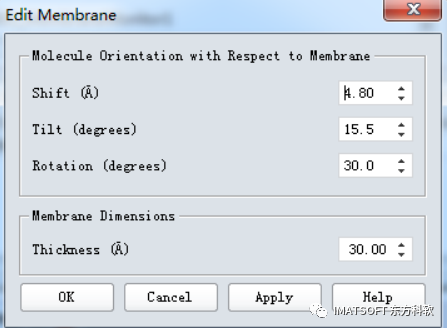
点击Modify…
打开Edit Membrane对话框。
设定Shift为4.8 Å,Tilt 为15.5度,Rotation 为30.0度。
点击OK。
点击3D窗口中任一位置,从而不选中蛋白质。
在菜单中Display Style中选择第一种表示方式.
调整后的膜结构中,胆固醇和棕榈酸分子的极性区都位于生物膜疏水区的外侧(图9)。
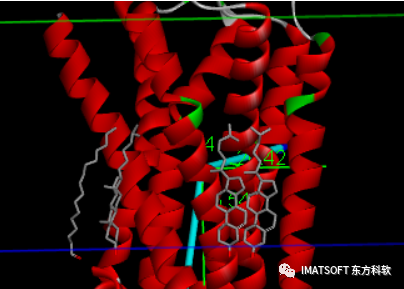
图9










 苏公网安备 32059002002276号
苏公网安备 32059002002276号
