

目的:通过此教程了解Discovery Studio软件中使用大量配体/大数据集中生成集合药效团
所需功能模块:Discovery Studio Client、Ensemble Pharmacophore Generation、Find Diverse Molecules、Common Feature Pharmacophore Generation、Ligand Profiler.
所需数据文件:akt1_actives.sd、kt1_decoys.sd
介绍
在药效团生成的早期,活性配体的数据集相对较少。近年来,ChEMBL等数据库提供了大量靶标的更多数据。
为了解决这个问题,集成药效团生成协议已经创建。该协议自动化并增强了公共特征药效团生成协议中使用的HipHop算法。它允许用户在比现有药效团生成协议所能管理的更大的数据集中快速识别配体簇,并且为每个簇生成一个药效团模型。综合起来,这些药效团模型形成了一个由不同假设组成的药效团,有效地代表了靶标。
本教程中的活性和非活性配体来自 Database of Useful Decoys: Enhanced (DUD-E) 的AKT1数据集。AKT1是三种密切相关的丝氨酸/苏氨酸蛋白激酶(AKT1, AKT2和AKT3)之一。AKT激酶调节许多过程,包括代谢、增殖、细胞存活、生长和血管生成。通过去除名称和结构重复的配体,制备了DUD-E配体集。为了缩短本教程的运行时间,在 Find Diverse Molecules的程序帮助下,从16400+ AKT1诱饵中选择了300个非活性配体的不同子集。
产生一个合成药效团
AKT1数据集中的293个活性配体传统上需要用户手动选择最多32个活性配体作为共同特征药效团生成协议的输入。集成药效团生成协议通过使用药效指纹对大型输入活动数据集进行聚类,并从每个聚类中选择前5个配体来生成共同特征药效团,从而实现自动化。每个簇的10个药效团用活性和非活性配体进行验证,以选择更佳假设来模拟该簇的化学型。
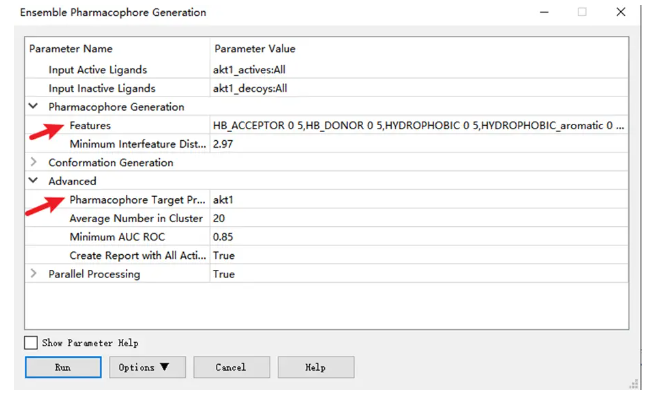
1. 打开 Samples | Tutorials | Pharmacophore | akt1_actives.sd 和 akt1_decoys.sd 文件。药效团特征添加HYDROPHOBIC_aliphatic 和 NEG_IONIZABLE,将药效团命名前缀改为akt1。如下
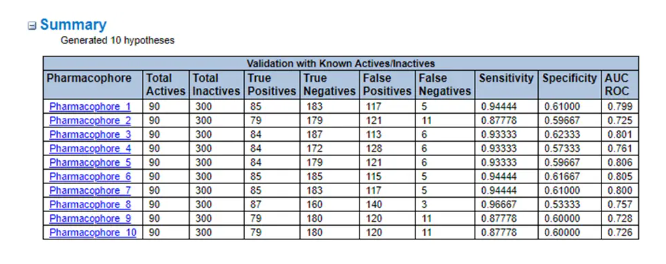
等待运行结束后,查看结果。这打开了显示簇和药效团验证的表格结果。在第一个Cluster active / inactive的Intra-Cluster Validation选项卡上,你可以看到每个Cluster的ROC评分都大于0.85,这是由最小AUC ROC参数设置的。该表包含了生成药效团的簇中每个药效团验证的混淆矩阵的细节。理想情况下,我们希望药效团模型具有高数量的真阳性和低数量的假阳性。Pharm 1映射了群集1中96个配体中的82个,而错误地映射了300个非活性配体中的1个。
将光标移到药效团1上,可以看到药效团的特征。
表中的Cutoff Score是药效团FitValue,基于平衡精度确定了活性配体与非活性配体的更佳区分:
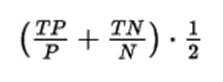
打开Validation with All Actives/Inactives选项卡。
从第一个标签的相同药效团的验证性能显示。然而,在这个表中,它们对293个原始活性配体的整个数据集的性能,而不仅仅是它们所产生的簇中的那些。该模型的Sensitivity指标下降了,但这并不令人惊讶,因为现在有更多来自不同簇的具有不同化学类型的活性配体。
打开Validation Failures选项卡。
如图所示,默认的10个共同特征药效团中没有一个生成模型,其ROC评分大于0.85。提供ROC评分和其他指标的目的是,如果药效团模型具有接近临界值的统计值,或以不同的方式符合您的项目标准,您仍然可以将其纳入您的目标药效团集合中。
这些验证失败的第一个簇包括90个活性配体,其中只有59个被检索到。这可能是因为选择的5个配体作为药物团阐释的先导不能很好地表征整个簇。您可以决定在此簇上尝试另一种选择方法,以生成代表该簇的化学类型的药效团模型。
簇6-15每个包含少于10个配体,正如预期的那样,它们没有产生药效团。此时,您可能想要合并一些失败的集群,并选择一个不同的代表性训练集来对它们建模。
挽救失败的集群
>在这一步中,你将从第一个失败的簇中提取配体,看看你是否可以选择不同的输入配体来生成药效团。您将使用自动方法选择5个配体,但也可以仔细阅读配体并为集群手动选择训练集。
点击SD 2打开包含第一个失败簇的90个配体的分子窗口。打开Find Diverse Molecules 按照如下设置,运行

此举选择了5个不同的配体。打开 Common Feature Pharmacophore Generation,按照如下参数设置并运行:

生成10个假设药效团。Summary部分显示了一个类似于Ensemble Pharmacophore Generation协议输出的验证表。共同特征药效团生成协议的结果与之前协议的主要区别在于,该集群返回10个模型而不是一个,并且产生更佳AUC的截止分数(FitValue)没有被纳入这些模型中。
通过分析该表,您可以根据药效团6的ROC评分选择药效团6作为更佳性能模型。
利用组合药效团进行虚拟筛选
你现在可以使用集成的AKT1药效团进行虚拟筛选。您将使用来自[scPDB](http://bioinfo-pharma.u-strasbg.fr/scPDB/)的配体数据库来识别AKT1的新的潜在引子。
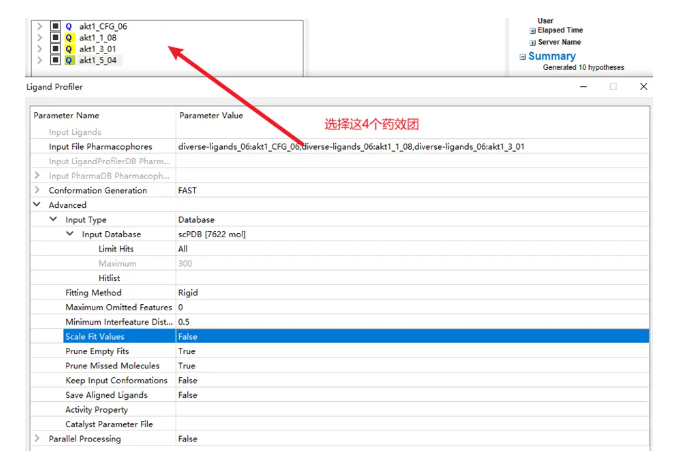
选择药效团6,接着locate刚刚运行好的Ensemble Pharmacophore Generationde 结果,然后扩展Cluster文件夹,选择所有的 ds_chm 文件(这些是成功的药效团),拖拽到药效团6下方。双击diverse-ligands_06改名为akt1_CFG_06。
打开Ligand Profiler,按如下参数进行设置并运行。
结果出来后,点击Fits Organized by Molecule来打开Fits by Molecule报告,单击FitValue列标题两次对表进行排序,来从数据库中寻找先导化合物。

许多最高分FitValue将与AKT1蛋白配体复合体相对应。然而,对于你的先导识别目标来说,更有趣的可能是其他尚未被称为AKT1结合配体的配体,它们可能被重新用作AKT1结合剂。









 苏公网安备 32059002002276号
苏公网安备 32059002002276号
