

目的:采用Discovery Studio,以一个蛋白为实例,示范定义受体结合位点的操作过程。
所需功能和模块:Discovery Studio Visualizer client
所需数据文件:1kim.pdb
所需时间:10分钟
介绍
受体结合位点的定义对于分子对接和打分都非常重要。寻找结合位点有两种方法:在受体空腔中寻找结合位点和在指定位置寻找结合位点。
本教程中,会介绍如何寻找并定义结合位点。具体包括:
• 基于蛋白空腔定义结合位点
• 基于指定位点定义结合位点
准备体系
在文件浏览器(Files Explorer)中,找到并双击打开Samples | Tutorials | Receptor-Ligand Interactions| 1kim.pdb。
在分子窗口中将打开一个蛋白质三维结构,该蛋白已经过一定的预处理。
在工具浏览器(Tools Explorer)中,展开Receptor-Ligand Interactions|Define and Edit Binding Site,点击Define Receptor,将选择的蛋白分子1kim定义为受体分子。(图1)

在受体空腔中寻找结合位点
在工具浏览器(Tools Explorer)中,展开Receptor-Ligand Interactions|Define and Edit Binding Site,点击From Receptor Cavities。
通过寻找的空腔来定义受体中可能的结合部位。
在系统视图(Ctrl+H)中展开1kim。
可以看到识别出9个可能的结合位点(Site1-9),更大的可能的结合位点被展示在图形视图中(图2)。

每一个找到的结合位点都由绿色的点(Binding Site)和一个红色的球(SBD_Site_Sphere)构成,绿色的点显示了空腔所在的位置和空腔形状,并被红色的球体所囊括。
红色的球体为SBD_Site_Sphere,可通过Receptor-Ligand Interactions | Define and Edit Binding Site工具面板下的Show/Hide Site Spheres选择显示与否。
在工具浏览器(Tools Explorer)中,展开Receptor-Ligand Interactions|Define and Edit Binding Site,点击

按钮可以观察不同的Site。
此外,可以通过Receptor-Ligand Interactions |Define and Edit Binding Site工具面板下, Expand或Contract按钮来调大或缩小binding site和SBD Site Sphere的大小。
在指定位置寻找结合位点
在系统视图(Ctrl+H)中展开1kim,将上述步骤产生的9个位点全部选中,然后点击键盘上的Delete予以删除。
在系统视图(Ctrl+H)中展开1kim的链A,选择残基HIS58和VAL70。
在工具浏览器(Tools Explorer)中,展开Receptor-Ligand Interactions|Define and Edit Binding Site,点击From Current Selection,在HIS58和VAL70处寻找可能的结合部位(图3)。
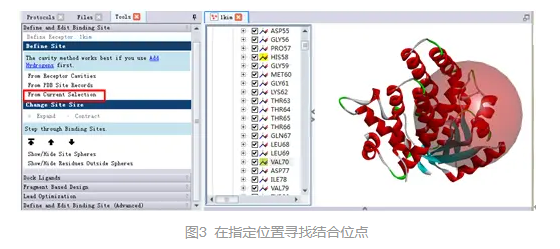
注:该方法也可以选择任意原子或体系来进行结合位点的定义,如复合物晶体结构中自带的小分子配体等。
修改活性部位球体半径
单击选中SBD_Site_Sphere球体,点击鼠标右键选择Attributes of SBD_Site_Sphere…,打开SphereObject Attributes对话框,在半径(Radius)选项输入设定的数值,点击OK按钮即可修改球体半径(图4)。










 苏公网安备 32059002002276号
苏公网安备 32059002002276号
